2. Epigenética
2. Epigenética
A diferencia de la genética, que estudia variaciones en la secuencia, la epigenética consiste en la expresión diferencial de los genes que tiene lugar sin cambios en la secuencia. En este sentido, podemos agrupar las alteraciones epigenéticas en la metilación del ADN, acetilación de histonas y metilación de histonas. (figura 1).
El ADN no metilado y las histonas con colas acetiladas permiten el ensamblaje de los factores de transcripción y de la actividad de la ARN polimerasa, indispensables para la transcripción . La acción de la ADN metiltransferasa y la histona desacetilasa hacen que se condense la cromatina, volviéndola inaccesible a los factores de transcripción.
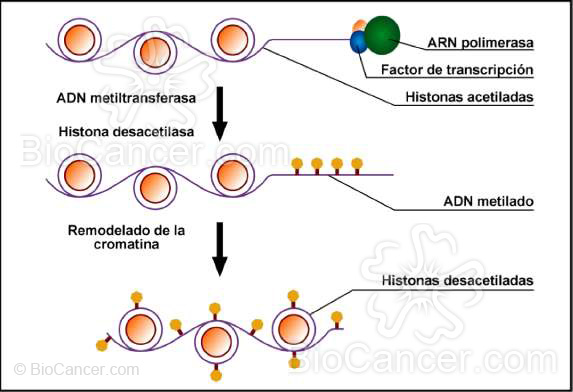
Figura 1.- Cambios epigenéticos
El cáncer puede ser considerado como una enfermedad epigenética. La carcinogénesis y la metilación aberrante del ADN están claramente relacionadas. Por ello, desde hace más de dos décadas, la patogénesis y progresión del cáncer se estudian también desde este punto de vista, ya que la combinación de cambios en este nivel se asocia con la inhibición de la transcripción de genes clave en la regulación celular, control del ciclo celular y apoptosis.
El gen BRCA1 es un claro ejemplo del efecto epigenético en los procesos cancerígenos. Se ha demostrado que:
Además, un estudio realizado en 2006 comparó los resultados clínicos de pacientes con cáncer de ovario con el gen BRCA1 silenciado por hipermetilación del promotor, pacientes con mutaciones en la línea germinal de BRCA1 y pacientes con genotipo salvaje. El intervalo libre de enfermedad y la supervivencia global fueron significativamente más cortos en pacientes con el promotor de BCRA1 hipermetilado (Chiang et al, 2006).
Estos datos sugieren que la metilación del promotor de BRCA1 está asociada a peor pronóstico y que BRCA1 podría formar parte de esquema global de genes metilados asociados a enfermedades agresivas (Chiang et al, 2006).
El carcinoma hepatocelular (HCC) es una de las patologías más comunes en todo el mundo. La patogénesis molecular de esta enfermedad se caracteriza por múltiples cambios genéticos y epigenéticos, como son mutaciones somáticas en el gen supresor de p53 (TP53) y activación de la señal de traducción de WNT (http://en.wikipedia.org/wiki/Wnt_signaling_pathway) . La exposición a aflatoxina B1 es uno de los factores de riesgo, ya que puede provocar transversiones (G:C a T:A) en el TP53 y favorecer que infecciones por hepatitis B causen mutaciones en p53. Así, la detección de TP53 mutado en plasma funciona como indicador de exposición a aflatoxina y de riesgo de HCC (Hussain et al, 2007).
La hipermetilación de GSTP1 es la alteración molecular más común en el cáncer de próstata. El grado de metilación de GSTP, detectado en orina, ha sido propuesto como marcador diagnóstico para mejorar la especificidad del test de PSA (Prostate-specific antigen) y ayudar a distinguir pacientes con cáncer prostático de aquellos con hiperplasia benigna, aunque aún es necesario un estudio prospectivo (Woodson et al, 2008).
El cáncer de pulmón es el de mayor índice de mortalidad debido en gran parte a la falta de métodos para una detección temprana. Presenta una serie compleja de cambios genéticos y epigenéticos que llevan al crecimiento celular descontrolado y a la metástasis. A diferencia de las alteraciones genéticas, las epigenéticas (metilación del ADN y acetilación de histonas) son eventos tempranos en la tumorogénesis pulmonar. Un estudio realizado combinando inhibidores farmacológicos de la metilación de ADN y la desacetilación de histonas en líneas celulares de cáncer de pulmón de células no pequeñas, reveló que los genes que codifican para neuronatina, metalotionina 3 y cistatina E/M aparecían frecuentemente hipermetilados y se expresaban menos. Otros cuatro genes (cilindromatosis, CD9, factor de activación de la transcripción 3 y oxitocina) eran regulados por la desacetilación de histonas y su expresión también era menor. Estos genes podrían ser considerados como supuestos reguladores del crecimiento celular en este tipo de neoplasia, así como biomarcadores epigenéticos (que podrían ser usados para estrategias de detección temprana) y dianas terapéuticas (Zhong et al, 2007).
Las investigaciones actuales combinan distintos tipos de agentes con la identificación de nuevas dianas dentro de la familia de enzimas epigenéticos (Allen, 2007):
