3.1 Transcriptoma
3.1 Transcriptoma
El transcriptoma se define como el conjunto de ARN mensajero resultante de la traducción del genoma bajo determinadas condiciones. Hay que tener cuenta que en algunas células la actividad transcripcional es constante durante toda su vida, pero en otras depende de estados fisiológicos o patológicos y estímulos específicos.
El splicing es el mecanismo post-transcripcional en el que se cortan los intrones y se acoplan los exones que codificarán las proteínas. Las mutaciones en las zonas de empalme, heredadas o adquiridas, suelen ser el motivo del splicing aberrante o patológico que produce un ARNm anómalo (Brinkman, 2004).
Este fenómeno se ha observado en genes implicados en carcinogénesis, como TP53, BRCA1, MLH1, FHIT o TSG101 (Melver et al, 2000). Por ello, el estudio del splicing y la caracterización de las variantes de éste adquieren importancia a la hora de entender las transformaciones neoplásicas.
Microarrays o chips son de gran utilidad para analizar simultáneamente un gran número de transcritos en un solo ensayo, comparar muestras de tejido sano y afecto, y extraer información del estado de la enfermedad a este nivel. Los patrones que caracterizan cada estado podrán ser aplicados a la práctica clínica.
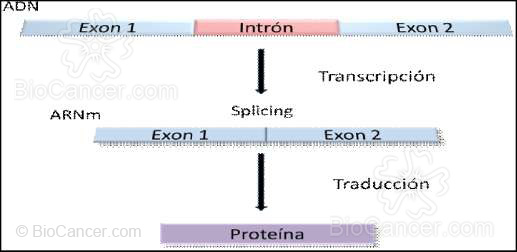
Figura 2.- Procesos de transcripción y traducción.
